Une IA qui éclaire le vivant : au CBIO décrypter la biologie pour mieux soigner

La recherche menée au CBIO s’inscrit à l’interface entre mathématiques, informatique, biologie et médecine. Sa mission est claire : concevoir des méthodes d’apprentissage automatique et d’IA capables de donner du sens aux données biologiques contemporaines, dont la quantité et la complexité ne cessent de croître.
Ces travaux répondent à trois objectifs scientifiques majeurs :
Pour atteindre ces objectifs, le CBIO s’appuie sur une équipe de chercheuses et chercheurs permanents aux expertises complémentaires : Chloé-Agathe Azencott, Éloïse Berson, Florian Massip, Vincent Mallet, Véronique Stoven et Thomas Walter. Le CBIO a un partenariat stratégique avec l’Institut Curie, centre de recherche et hôpital dédié au cancer, et est affilié à l’unité « Oncologie Computationnelle », ce qui lui permet de s’attaquer à des problèmes complexes et de répondre à de besoins cliniques concrets en oncologie.
Être capable de prédire l’évolution d’une maladie ou l’efficacité d’un traitement est aujourd’hui un enjeu majeur en oncologie. L’IA offre des méthodes puissantes pour intégrer de multiples sources de données et produire ce type de prédictions. Un obstacle central pour ces approches tient toutefois à la nature même des données analysées. On parle de données en très haute dimension lorsque l’on mesure un très grand nombre de variables pour un nombre limité de patients ou d’échantillons.
Par exemple, dans les études génomiques, l’objectif est d’identifier des liens statistiques entre des mutations – souvent au nombre de plusieurs millions – et un phénotype clinique. En transcriptomique, on analyse l’expression d’environ 20 000 gènes. Dans les deux cas, le nombre de patients à partir desquels les modèles d’IA peuvent apprendre est typiquement plusieurs ordres de grandeur inférieur au nombre de variables mesurées, ce qui rend les méthodes d’analyse classiques fragiles et parfois trompeuses.
Le CBIO adopte alors plusieurs stratégies complémentaires pour répondre à ce problème :
Ces approches permettent non seulement de prédire, mais surtout d’identifier les mécanismes biologiques réellement impliqués, un enjeu clé pour la recherche biomédicale.
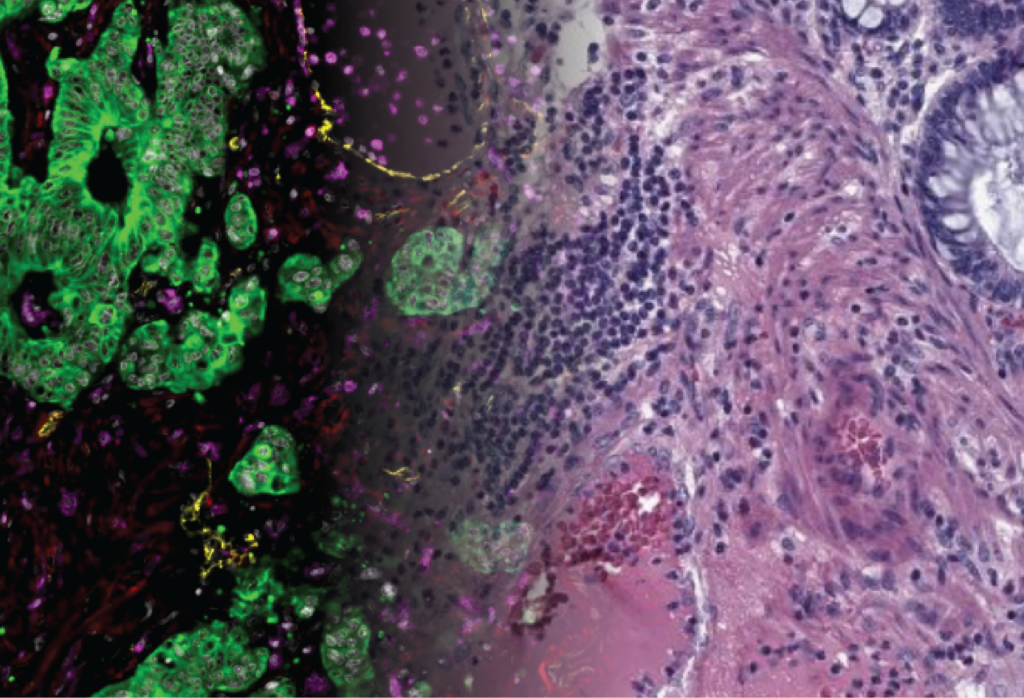
Parmi les axes de recherche les plus visibles du CBIO figure la pathologie computationnelle, qui exploite l’IA pour analyser des images de tissus biologiques issues de l’histopathologie, discipline destinée au diagnostic par l’étude microscopique des tissus. Ces images, obtenues à partir de coupes de tissus colorées et observées au microscope, sont aujourd’hui numérisées sous forme de lames entières (whole-slide images) pouvant atteindre plusieurs gigaoctets.
L’enjeu : extraire automatiquement des informations moléculaires ou cliniques à partir de la seule morphologie des tissus.
Les chercheurs du CBIO ont développé des méthodes capables de :
Concrètement, les images sont découpées en milliers de petites régions, analysées par des modèles d’IA pré-entraînés sur des millions d’images médicales, puis agrégées pour produire une prédiction à l’échelle du patient. Ces travaux ouvrent la voie à une oncologie plus rapide, moins coûteuse et plus accessible, sans recourir systématiquement à des analyses moléculaires lourdes. Dans l’ensemble de ces travaux, le CBIO collabore étroitement avec des services d’anatomopathologie hospitaliers, notamment à l’Institut Curie avec Anne Vincent Salomon, directrice de l’IHU Cancers des Femmes (Institut Curie – Universitéte PSL – Inserm) et médecin pathologiste cheffe du Pôle de Médecine diagnostique et théranostique (PMDT), ainsi qu’Yves Allory, Chef de l’équipe de recherche « Oncologie moléculaire » au sein de l’Unité Biologie cellulaire et Cancer (UMR144) et Chef du service Pathologie.
Une autre avancée majeure concerne la transcriptomique spatiale, une technologie récente qui permet d’observer non seulement quels gènes sont exprimés dans un tissu, mais aussi où ils le sont précisément. Concrètement, cette approche associe à chaque point d’un tissu une mesure de l’activité de milliers de gènes, parfois jusqu’à 20 000 gènes à la fois, tout en conservant leur position exacte dans l’espace. Elle offre ainsi une vision fine de l’architecture d’un tissu, en reliant la structure du tissu à son fonctionnement moléculaire.
Ces données sont particulièrement riches, mais elles sont aussi complexes et coûteuses à produire et à analyser. Elles soulèvent de nouveaux défis informatiques, comme la déconvolution cellulaire, qui consiste à déterminer quelles cellules contribuent à un signal mesuré sur une zone donnée, ou encore l’intégration de plusieurs types de données, par exemple des images de tissus et des mesures moléculaires. Un autre enjeu consiste à établir des liens prédictifs entre différentes modalités, afin de compléter une information par une autre.
Pour répondre à ces défis, les chercheurs du CBIO et de l’équipe de Biologie des Systèmes de l’Institut Curie dirigée par Emmanuel Barillot ont développé des modèles d’IA capables de prédire l’expression des gènes à l’échelle de la cellule à partir de l’aspect visuel d’un tissu observé au microscope. Ces modèles peuvent estimer des informations moléculaires très détaillées à partir d’examens faits en routine clinique, sans avoir recours systématiquement à des technologies lourdes et coûteuses.
Ces approches ouvrent des perspectives vers une biologie spatiale intégrative combinant images, données génétiques et informations cliniques. Elles sont particulièrement prometteuses pour mieux comprendre des maladies complexes comme le cancer, où l’organisation spatiale des cellules joue un rôle clé dans l’évolution de la maladie et la réponse aux traitements.

Les recherches du CBIO couvrent également la biologie structurale, qui s’intéresse à la forme tridimensionnelle des protéines et des ARN. Grâce à l’IA, il devient possible de :
Parallèlement, le centre développe des méthodes pour réconcilier les modèles statistiques de l’IA avec les modèles mécanistiques de la biologie, afin de passer de la corrélation à la compréhension des mécanismes biologiques sous-jacents.
Ces travaux ont été mis en lumière lors du Workshop IA organisé en décembre 2025 à Mines Paris – PSL. Pensé comme un moment d’échange interne, l’événement a permis aux enseignants-chercheurs, doctorants et ingénieurs de présenter leurs projets, outils et plateformes, à travers des présentations orales et des posters.
Au-delà de la diversité des sujets, le workshop a mis en évidence une dynamique commune : construire une IA ancrée dans le réel, de s’intégrer dans des systèmes complexes et de s’attaquer à des enjeux sociétaux majeurs
En développant une IA robuste, explicable et ancrée dans la réalité biologique, le CBIO contribue à transformer la manière dont la science du vivant est étudiée et appliquée. Entre recherche fondamentale, découverte de traitements et médecine de précision, ses travaux illustrent comment l’IA peut devenir un outil de compréhension du vivant, au service d’innovations médicales concrètes et responsables.

Comprendre, prédire et optimiser le comportement des matériaux est un défi central pour l’industrie, de l’aéronautique à l’énergie, en passant par les...